作为近年来快速发展的新兴技术,空间转录组学已经极大地改变了生物和医学多个领域的研究范式。这一技术保留了复杂组织中细胞的空间定位信息,并且以多细胞团、单细胞或亚细胞分辨率进行转录组分析。细胞空间定位信息与其分子特征谱的耦合产生了新型的多模态高通量数据资源,这对高效的数据分析与信息挖掘方法的开发提出了新的挑战。由于生理组织样本的复杂性与技术的局限性,空间转录组数据具有高度的非理想性。其数据结构复杂、信噪比低、稀疏性强且覆盖度不均一,这给数据的深度分析和生物学信息的解析带来了一系列挑战。此外,多个技术路线的空间转录组学正在快速迭代发展,需要现有数据分析理论的发展和创新技术工具的开发,从而充分发挥这一变革性技术的潜力。
近年来,针对空间转录组数据各个维度的深度解析与信息挖掘需求,生物信息方法学研究非常活跃,同时也面临诸多挑战。2024年3月25日,金沙js4399首页生命学院杨雪瑞课题组应邀在WIREs RNA发表了题为“探索空间转录组景观:计算方法如何导航”(Navigating the landscapes of spatial transcriptomics: How computational methods guide the way)的长篇综述文章,系统总结了当前空间转录组数据的常用解析方法,针对不同角度与层面的分析任务和信息挖掘需求,讨论了现有方法的设计思路与潜在的局限性,并就未来相关方法学理论与设计的方向和策略,提出了自己的观点。
空间转录组分析有不同的技术路线,当前的主流技术主要基于高通量测序或高分辨成像。总的来说,空间转录组数据的分析包括不同技术路线的数据预处理、生物学信息的挖掘和空间解析度下的知识重建(图1)。本综述详细总结了针对这一流程中各分析任务的生物信息方法,包括空间反卷积(图1A)、基因插补(图1B)、图像对齐和细胞分割(图1C)等数据预处理方法,以及空间基因表达模式的鉴定、空间数据的整合(图1D)、空间聚类(图1E)、基于scRNA-seq数据的空间重建(图1F)、细胞互作的推断(图1G)等深度挖掘方法。此外,本综述还深入讨论了空间数据的模拟方法,这是一个在近一年内迅速发展的新领域。
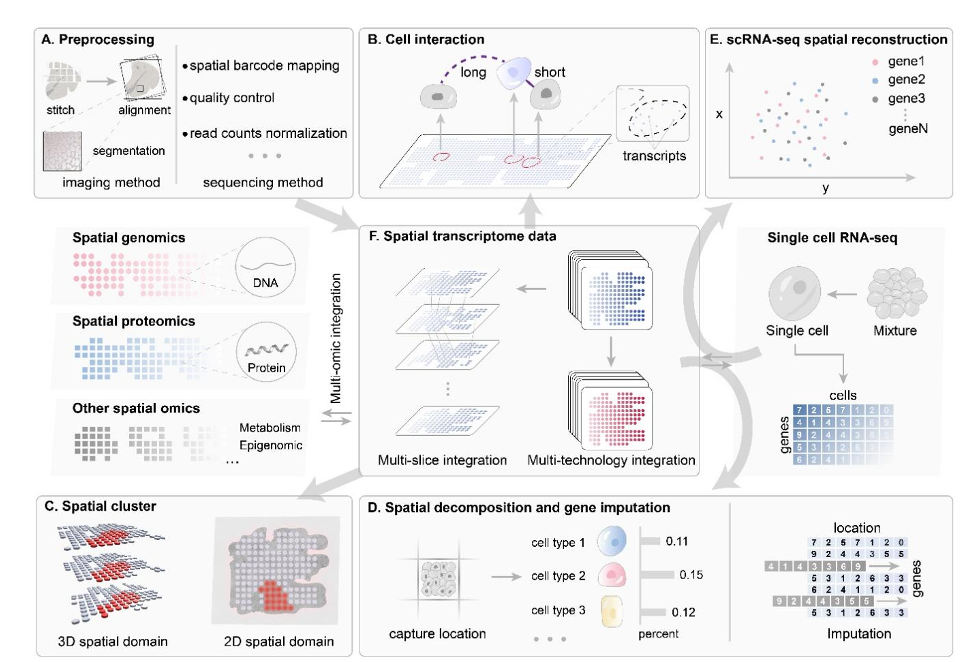
图1. 空间转录组数据的深度分析任务
在以上分析任务中,全面鉴定细胞间的复杂互作对于组织功能、发育和复杂疾病的研究至关重要。此前推断细胞互作的方法通常基于先验性信息所设计。此类方法容易受到不同空间区域不均衡特征谱分布的影响,并且强烈依赖配-受体对的先验知识或预定义的特征(如细胞类型信息)。此外,由于单细胞空间转录组数据通常具有缺失值多、信噪比低等问题,大多数细胞的配-受体表达信息往往残缺不全,这对于全面识别细胞互作关系具有很大的挑战性。针对该信息挖掘需求,杨雪瑞课题组此前开发了DeepLinc工具。该方法不依赖于先验的配-受体信息,使用基于深度学习的AI框架,从空间邻近细胞学习影响细胞互作的转录组隐藏特征,从头重建完整的细胞互作网络(Genome Biology,2022)。
在本次发表的综述论文中,作者重点剖析了未来的方法学研究需要解决的潜在挑战。随着空间转录组分析技术的快速发展,数据的复杂性和维度也在不断增加(图2),其中包括:从2D到3D数据的扩展;揭示组织内时空动态的时空组学分析;多模态和多组学数据的可扩展和可解释性分析;高解析度分子特征的空间插补以及亚细胞解析度的分子互作信息挖掘等。为解决这一系列挑战,未来的方法学研究需要设计理论的创新以及更高解析力的分析技术,特别是高性能AI技术的引入。
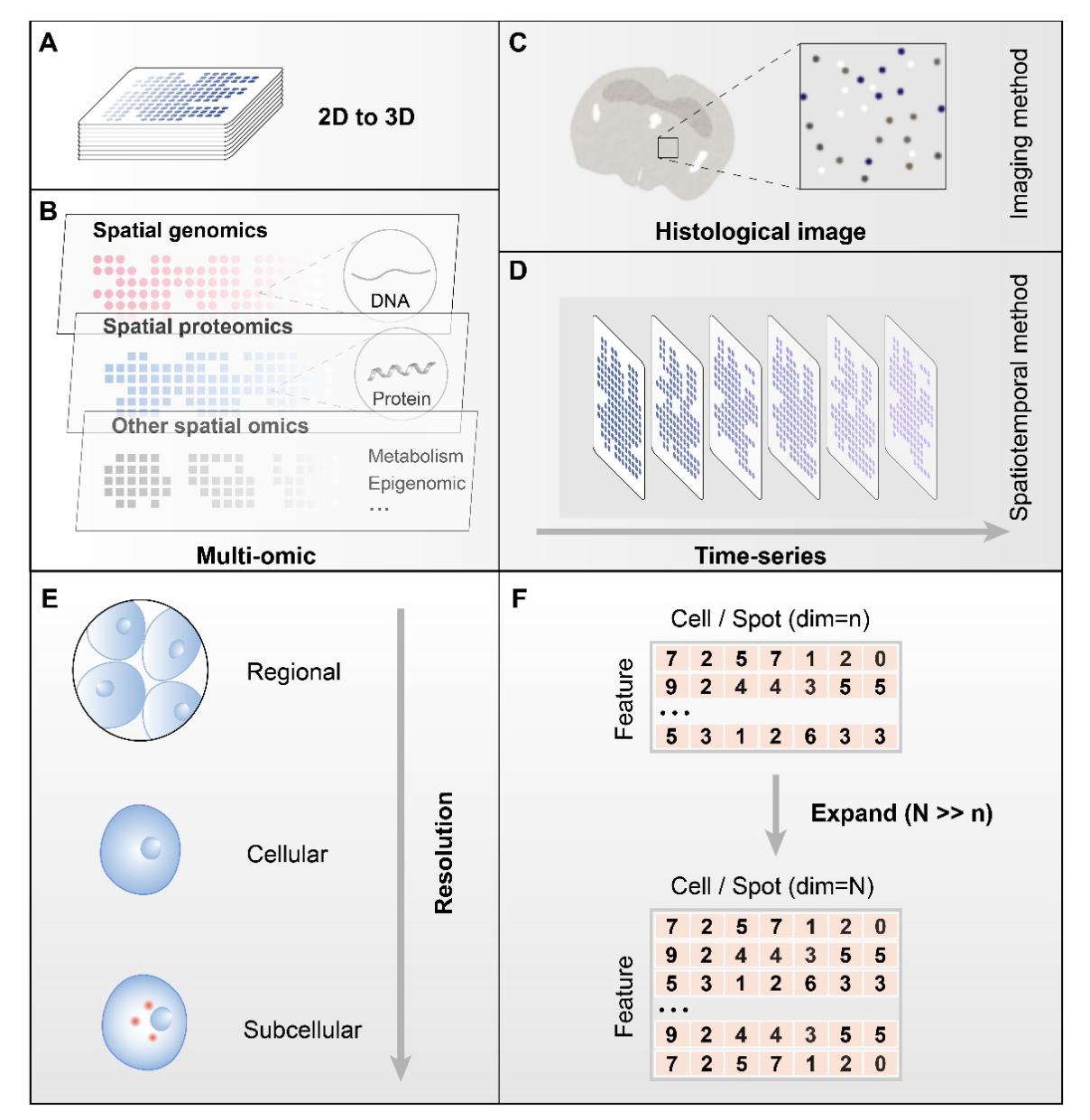
图2. 空间转录组数据复杂性和特征维度的增加
通过本综述文章,作者对更高水平的生物信息方法学研究提出展望,包括更强大、更灵活的分析框架,更高效的数据和信息集成策略,以达到更高的预测效率和准确性,并更深入地考虑数据和生物过程的复杂性。作者特别强调了知识迁移促进新的生物学发现的观点:与空间转录组数据的独立分析相比,与其他数据或知识源(例如基因组、蛋白质组、临床数据和分子调控网络等)之间的知识迁移策略更有利于全面解析生物系统的复杂性,以进一步发现新的空间模式、空间调控基因、空间编码细胞过程的分子途径和功能模块。
总而言之,空间转录组学技术为理解各类生理或病理组织的形成与功能提供了丰富的信息,而实验和计算方法的协同进步能够充分发挥空间转录组学的潜力。本综述全面、系统地剖析了空间转录组学面临的分析任务,为领域内研究人员提供最新的参考,并进一步激发新的数据分析理论、模型和算法的讨论和开发。生命学院杨雪瑞课题组博士后李润泽与2022级博士生陈旭为论文的共同第一作者,杨雪瑞与李润泽为论文的共同通讯作者。该工作得到了国家重点研发计划、国家自然科学基金、金沙js4399首页自主科研计划的资助。
原文链接:https://wires.onlinelibrary.wiley.com/doi/10.1002/wrna.1839